端粒酶是负责端粒维持的核糖核蛋白(RNP),具有复杂的生命。复杂是因为它是由多种蛋白质和RNA组成的,复杂是因为它经历了许多变化,并通过不同的细胞区室。因此,人们开发了许多方法来发现端粒酶的成分,深入了解其结构和功能,并弄清楚端粒酶生物学最终与人类健康和疾病的关系。虽然一些旧的黄金标准方法仍然是确定端粒长度和测量端粒酶活性的关键,但新技术正在提供有希望的新方法来获得我们以前从未接触过的详细信息。因此,我们认为及时地回顾一下已经揭示端粒酶RNP信息的方法,并概述一些可以使用新方法回答的剩余问题。
在过去的90年里,理解染色体生物学的方法发生了巨大的变化。对于染色体的末端,Muller在1938年创造了“端粒”,通过哲学和生物化学方法来理解它们的生物学相关性,有助于我们对染色体生物学、细胞衰老、癌症和端粒相关疾病的全面了解。由于多年来发展了许多强有力的方法,我们现在对端粒是如何维持的以及所涉及的过程如何影响健康和疾病有了相对较好的了解,但仍然存在许多问题。Muller最初将染色体末端描述为重复的或无活性的“基因”(基因指的是核酸块,而不是我们今天所知道的基因),这些基因对x射线损伤具有抵抗力(Muller 1938)。与此同时,芭芭拉·麦克林托克(Barbara McClintock)的工作证明了染色体末端的特殊性质,它显然能够在断裂后愈合(麦克林托克1941)。然而,当涉及到修复时,任何类型的末端都是有问题的,Olovnikov(1971)的初始思想实验(也称为Gedankenexperimente)讨论了“边缘切断术”理论:染色体末端有害损失的可能性。在那篇论文中,人们推测这种损失应该被克服,至少在某些细胞类型中,因此端粒可能被一种假设的未知酶延长。此后不久,James Watson(1972)基于对T7噬菌体DNA的研究,描述了染色体末端的不完全复制,也暗示了端粒延伸的潜在需求。由于这些对遗传、哲学和生物化学实验的令人兴奋的初步解释,一系列的方法帮助揭示了与端粒维持有关的蛋白质、RNA和细胞过程。端粒酶是负责端粒延伸的酶,为了最终发现端粒酶并对其进行鉴定,需要许多不同的方法来鉴定构成这种核糖核蛋白(RNP)的蛋白质和RNA,并测量其催化活性。在这篇综述中,我们讨论了用于研究端粒酶如何维持端粒的主要技术,包括用于研究DNA, RNA,蛋白质,全细胞/生物体和计算机方法的方法,重点是酵母和人类研究,并提到了纤毛虫的基础工作。
端粒由重复的DNA束组成,与结合的端粒蛋白一起,充当染色体末端的保护帽(Wellinger and Zakian 2012;Bonnell et al. 2021;林和切赫2021)。DNA复制机制无法在整个染色体长度上复制两条DNA链,导致端粒缩短和富含g的3 '悬垂(Wellinger and Zakian 2012;de Lange 2018)。在端粒酶阳性的细胞(如人类干细胞、纤毛虫和酵母)中,端粒可以延长并因此得以维持,而在端粒酶阴性的细胞(如人类体细胞)中,端粒不会延长,最终细胞进入复制性衰老。自从最初发现端粒和端粒酶以来,已经有许多方法在分子水平上研究端粒。首先,端粒长度测量(即测量端粒末端重复序列组成的DNA的长度)对于理解端粒酶如何维持端粒至关重要。两种主要方法,Southern blotting (Southern 1975;布莱克本和加尔1978)和荧光原位杂交(FISH;Lansdorp et al. 1996)提供了端粒总长度的测量,而Southern印迹法几十年来一直是金标准方法。这是端粒长度分析的第一种方法,起源于对纤毛虫的初步研究(Blackburn and Gall 1978;Blackburn和Challoner 1984),并且需要最少的培训,并借助于商业工具包的可用性。最初,该方法需要用Bal31消化分析DNA, Bal31是一种核酸酶,可以从末端降解dsDNA,从而缩短Southern blots中的末端限制性内切片段(TRFs),这明确表明重复束是末端DNA (Blackburn and Challoner 1984;Shampay et al. 1984;Walmsley和Petes 1985)。对于出芽酵母,单个端粒上的端粒重复束通常相对较短(~ 325 bp),通过使用独特的亚端粒探针,Southern blotting也可用于测量特定个体染色体末端的DNA重复长度。这样的测量产生了这样的结论:所有出芽的酵母端粒都携带大约325 bp(±50 bp)的端粒重复序列(Walmsley et al. 1984;沃姆斯利和皮特,1985),尽管观察到至少3号染色体上的左端粒更长,不符合这个想法(巴顿和阿斯特尔,1986)。在来自人类细胞的DNA中,通过Southern TRF法进行的大量端粒测量可以揭示端粒是否普遍受到细胞类型来源或培养条件的影响,但不能提供单个染色体末端的具体细节。由于人类端粒的长度和变异(5-15 kbp;de Lange et al. 1990;Canela et al. 2007), TRF条带会产生很长的涂片,这使得很难辨别大块端粒长度的细微变化。然而,定量FISH(荧光原位杂交)是一种信息丰富的替代方法,因为人类细胞染色体大小和端粒长度可以通过荧光强度识别和量化(Lansdorp等人,1996;有关详细审查,请参阅Lai et al. 2018)。尽管FISH可以在人类细胞中提供比Southern印迹法更多的细节,但它仅限于有丝分裂的细胞类型,并且需要显微镜方面的专业知识。然而,FISH也与流式细胞术(flow-FISH)相结合,可以快速得出端粒长度的结果,并能够用于临床应用(Baerlocher and Lansdorp 2003;Baerlocher et al. 2006)。flow-FISH的优点包括高通量分析的可用性和相对快速的结果,尽管它通常只用于一小部分细胞类型。也有基于PCR的人类端粒长度测量方法,如定量PCR (qPCR)和单端粒长度分析(STELA),可用于批量或单个端粒分析。qPCR和STELA具有成本效益高、获得结果速度快、输入DNA浓度低的优点(Baird et al. 2003;2002年,2009年)。然而,qPCR和STELA在技术和生物重复中都可能存在变异性(Aubert et al. 2012;Aviv et al. 2011),并且不能轻易地在研究之间进行比较。就STELA而言,迄今为止,它只能应用于具有独特亚端粒区域的少数染色体或小于8 kb的端粒(Aubert et al. 2012)。考虑到qPCR和STELA可能带来的技术挑战,它是否适用于广泛的应用以及所获得的信息是否超过TRF分析仍然值得怀疑。
尽管Southern blotting和FISH对端粒总长度具有稳健性和可重复性,但两者都不能提供所有单个端粒的特定碱基对长度测定,并且可能无法检测缺失的端粒。为了解决这些问题,长读测序技术(PacBio;Eid et al. 2009或Oxford Nanopore测序;Jain et al. 2016)正在被用来确定酵母(Sholes et al. 2022)和人类(Tham et al. 2023)的个体端粒长度。与Southern blotting和FISH方案不同,测序可以更容易地应用于大样本量,并已成功用于确定1000多个分离株的酵母端粒长度(D’angiolo等,2023)。与STELA或flow-FISH等其他方法不同,端粒测序还可以用于更大范围的细胞类型和组织,克服了有关细胞分裂状态或样品类型的一些限制。虽然这种新的端粒测序方法非常有前途,但仍然存在重复端粒序列比对问题,以及与个体性和人工制品有关的一般问题。尽管对个体端粒进行测序对人类端粒相关疾病的诊断具有巨大的潜力,但仍需要大量工作来创建和实施强大,高效和用户友好的DNA提取,测序和分析方案,以广泛使用和各种细胞类型。一旦克服了这些挑战,测序可能能够提供关于不同细胞类型的端粒长度模式或通过祖先预测端粒相关疾病和癌症概率的新信息。端粒测序还可以用来发现为什么一些染色体末端更长(它是否与特定基因的位置有关,例如交配型位点或必需基因?),特定端粒长度是否受到年龄或一般健康状况的影响,甚至可能用于发现核苷酸水平的基因组变化,这些变化可能会增加端粒酶对端粒的吸引力(图1)。
图1
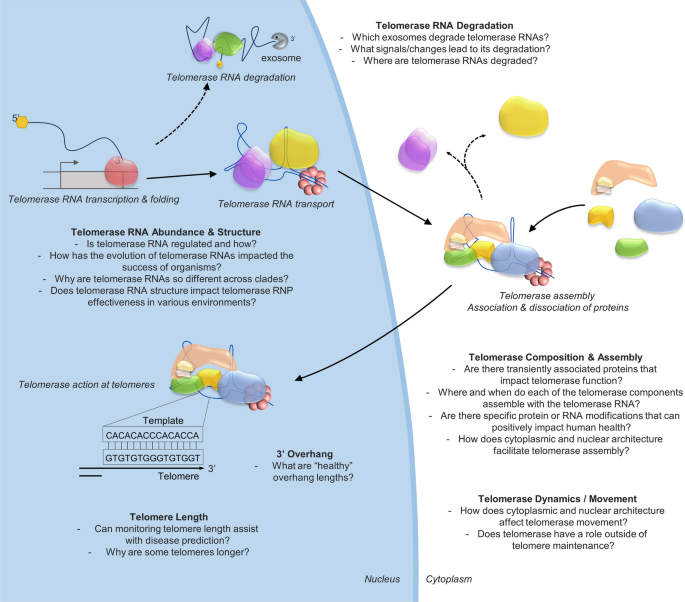
端粒酶生命周期的广义模型和一些有待回答的问题。这些问题列在以下小标题下:端粒酶RNA丰度和结构;端粒酶RNA降解;端粒酶的组成与组装;端粒酶动力学/运动;3 '悬垂和端粒长度。该模型基于酿酒酵母端粒酶过程,包括端粒酶RNP过程的一些物种特异性变异
除了端粒长度,早期有一个重要的观察,即端粒留下了一个3 ' '的单链悬垂(也被称为g尾;Klobutcher et al. 1981;亨德森和布莱克本1989;Wellinger et al. 1992)。这是端粒酶结合和延长端粒所必需的,在端粒酶亚基缺乏的人类细胞中,悬垂长度受到影响(Chai et al. 2006)。为了分析g -tail,开发了一种凝胶内杂交试验,可用于检测和量化单链端粒悬垂(Dionne和Wellinger 1996)。虽然这种方法需要一定的专业知识,但它无疑为研究3 ' g -tail提供了一种清晰的方法,并阐明了不依赖端粒酶的3 '端粒悬垂的产生以及端粒酶介导的端粒延长的时间(Wellinger et al. 1993;Dionne and Wellinger 1996)。凝胶现在被标准地用于人类细胞系中端粒延长(ALT)机制的表征,并且仍然是单链分析的金标准。后来,基于连接的方法,如连接介导的引物延伸(Jacob et al. 2001)和端粒重复寡核苷酸连接测定(T-OLA;Cimino-Reale等人(2001)提供了更精确的纤毛虫和人类细胞中3 '悬垂的测量,有助于确定短g尾(小于90 nt)的频率,这被电子显微镜等其他方法低估了。双链特异性核酸酶(DSN)方法也被开发用于检测长度至少为12个核苷酸的短悬垂(Zhao et al. 2008)。这种方法已经成功地用于人类细胞系,并提供了一种专门评估端粒酶功能障碍对悬垂长度的影响的方法。
所有的g尾分析方法都是劳动密集型的,因此不适合高通量分析。尽管3 '悬垂分析的复杂性和密集性,但它们并不局限于某些细胞类型,因此可以用于研究许多疾病表型或用于理解健康衰老,可能会回答:在每个生命阶段,什么样的g尾长度被认为是“健康的”?(图1)测序可能是大规模分析3 '悬垂的另一种方法,因为长读技术提供了对染色体末端的测序,并且随着测序质量和生物信息学工具的改进,两条同源链可以特异性对齐。执行这样的链特异性测序读数比对可以详细说明悬垂长度和总端粒长度,从而为任何和每种细胞类型提供端粒指标的全面视图。最终,这可能有助于个体和细胞类型特异性的医学分析和治疗。但是,在端粒悬垂的高通量方法被设计出来之前,仍然存在有关3 '悬垂如何与端粒紊乱有关的问题,以及悬垂长度对一般健康的总体影响。
端粒的最初发现自然导致了端粒重复添加过程的理论。生物化学有助于发现端粒重复加成是一个酶促过程;值得获得诺贝尔奖的发现(Greider and Blackburn 1985,1987)。一旦弄清楚端粒重复是由一种被称为端粒酶的逆转录酶类型维持的,那么分析端粒酶生物学的各个方面,从它的产生到消失,就变得至关重要了。评估催化端粒酶在各种情况下的活性,获得了端粒酶功能所需的蛋白质和RNA的特定信息。在酵母和人类细胞中,端粒酶经过一定的富集后,这种催化活性可以使用直接放射性核苷酸添加试验的适应性来测量(Morin 1989;Cohn and Blackburn 1995),最初是为纤毛细胞提取物而创建的(Greider and Blackburn 1985, 1987)。为了更灵敏的检测,后来开发了基于pcr的端粒酶重复扩增协议(TRAP)测定法(Kim et al. 1994)。TRAP检测方法已被修改为使用非放射性荧光标记的核苷酸以提高安全性和可及性(Herbert等人,2006年),或者包括扩增步骤以优化不同细胞类型(Blasco等人,1997年)。由于检测端粒酶活性仍然非常有用,现在有商业试剂盒使这项技术可用于临床。虽然上述针对酶活性的检测有助于直接识别端粒酶所需的蛋白质,但它们并不能说明端粒的组装或体内作用。端粒酶测定可用于单个细胞类型;然而,端粒酶的分离和纯化可能是困难的,因此不适合大规模应用。
虽然传统的端粒酶测定确实提供了关于整个细胞提取物的大量端粒酶活性的信息,但最近测量端粒酶活性的方法可以在单分子水平上评估RNP。这种程度的细节对于确定可能以特定方式影响端粒酶活性的因素是必要的:例如底物结合,核苷酸附加性和重复附加性。利用光学镊子对端粒酶在端粒底物上的催化作用进行了令人难以置信的详细观察(Patrick et al. 2020)。光学镊子提供迄今为止最高的分辨率,是一种体外方法,可以测试单个RNPs执行端粒重复延伸的能力。这些方法提供了与经典TRAP和放射性核苷酸加成法相同的信息,端粒酶的催化活性,但也证实了人类端粒酶的二级DNA结合位点,以及核苷酸逐步加成过程的核苷酸尺度细节(Patrick et al. 2020)。如此详细地了解端粒酶的活性,可以深入了解在核苷酸或重复过程中发生的端粒酶功能障碍,并提供有关某些端粒病变的更多信息。正如Opresko和Shay(2017)所讨论的那样,随着端粒维持临床相关测量的新工具的出现,端粒相关综合征将继续被发现和诊断。
端粒酶的一个重要组成部分是提供端粒延伸序列的模板RNA。因此,许多方法被用来测量端粒酶RNA的丰度、形状、加工和细胞内的位置。端粒酶RNA丰度特别有趣,因为RNA是酵母端粒酶的限制因素(Mozdy和Cech 2006;Bajon等人,2015),并在人体细胞中保持稳定的平衡(Xi和Cech, 2014)。此外,较高水平的端粒酶RNA与两种人类的异常效应有关(与癌症有关;Pruzan et al. 1990;Avilion et al. 1996)和酵母细胞(导致端粒缩短;Singer and Gottschling 1994),表明端粒酶RNA的浓度需要受到限制,才能保证健康的细胞功能。因此,考虑端粒酶rna的分析方法似乎是值得的。
人类和酵母的端粒酶rna已经通过Northern blot和qPCR成功定量,揭示在任何时候只有相对较少的分子(Avilion et al. 1996;Lingner et al. 1997b;莫兹迪和切赫2006;No?l et al. 2012;Tseng et al. 2015)。除了这些检测之外,单分子FISH已被用于端粒酶RNA丰度的“基本事实”测量,同时也提供了亚细胞定位信息(Zhu等人,2004;Gallardo et al. 2008;Bajon et al. 2015;Vasianovich et al. 2020)。使用荧光共振能量转移(FRET),精确测量细胞中人类端粒酶RNA丰度,除了用于端粒酶RNA与蛋白质或端粒之间的近距离相互作用外,还可以提供FISH的替代方法(Zhang等人,2022)。总的来说,这些分析有助于我们理解维持端粒酶RNA丰度对端粒酶功能和整体细胞健康至关重要。
在RNA的二级结构方面,使用系统遗传学的排列和折叠算法最初用于预测纤毛虫端粒酶RNA的结构(Romero和Blackburn 1991;McCormick-Graham and Romero 1995)和后来的脊椎动物,包括人类端粒酶RNA (Chen et al. 2000;Chen and Greider 2004)。酵母系统发育预测(Zappulla and Cech 2004;Dandjinou et al. 2004)通过位点特异性RNAse H裂解测定(Dandjinou et al. 2004)和二甲基硫酸盐敏感性测定(F?rstemann and Lingner 2005)在实验上得到了支持。引物延伸分析后选择性2 ' -羟基酰化(SHAPE;Wilkinson et al. 2006;Laterreur et al. 2018)有助于提高酵母端粒酶RNA某些区域的二级结构分辨率。这些二级结构使我们更深入地了解端粒酶rna在不同王国之间的差异,并为理解端粒酶RNP的整体结构提供了一个起点。更具体的细节已经通过核磁共振(NMR)获得,它揭示了端粒酶RNA伪结的折叠(Theimer et al. 2005)。
除了对RNA结构的总体预测之外,northern blot和qPCR等方法还揭示了端粒酶RNA更长的未加工形式的存在(Seto et al. 1999;No?l et al. 2012;Tseng et al. 2015)利用cDNA末端3 '快速扩增(RACE)结合深度测序进一步鉴定了人类端粒酶rna 3 '末端的异质性(Goldfarb and Cech 2013)。对于5 '端,TMG-RNA免疫沉淀和抗tmg免疫印迹显示在人和酵母端粒酶rna上有三甲基鸟苷帽(Seto et al. 1999;Jády et al. 2004)。
然而,考虑到细胞群体中端粒酶RNA数量的广泛分布,关于端粒酶RNA水平是否在整个细胞周期中波动的信息仍然缺失(图1)(Vasianovich et al. 2020)。这可以通过活细胞显微镜和单细胞的长期观察来回答,通过使用微流体设备与荧光RNA适配体和蛋白质(稍后讨论)相结合而成为可能。准确、快速和详细的端粒酶RNA丰度测量可以帮助我们全面了解衰老,当与端粒长度分析和端粒酶活性相结合时,可以帮助管理健康的衰老或预测组织特异性疾病的发作。
除了端粒酶RNA丰度外,端粒酶RNA的半衰期也用转录抑制法测定。该方法在人类细胞(Yi et al. 1999)和酵母细胞(Larose et al. 2007)中发现了端粒酶RNA异乎寻常的长稳定性,然而该方法最终会杀死细胞,并且在酵母端粒酶RNA的情况下,它只能提供> 1小时的半衰期估计(Larose et al. 2007)。到目前为止,还没有进一步的方法被用来获得酵母和人类端粒酶RNA的更具体的半衰期信息,但弄清楚这种寿命的原因可以为端粒酶的生物发生提供宝贵的信息,并为治疗端粒酶RNA疾病提供新的途径。还有一个问题:端粒酶RNA降解的机制是什么?(图1)使用基因技术,可以选择性地靶向“老”端粒酶rna而不影响细胞功能,将有助于研究这些长寿的rna。像Dendra2这样的可光切换荧光蛋白(Gurskaya et al. 2006;Auboiron et al. 2021)可与rna -蛋白质结合系统(即ms2 -茎环:ms2 -外壳蛋白结合;Bertrand et al. 1998;Gallardo et al. 2008;Laprade et al. 2020)或通过诱导RNA修饰(如Vasianovich et al.(2020)),实现对旧端粒酶RNA的特异性和长期分析。另一方面,端粒酶RNA降解可以通过删除靶向RNA的外切酶或使用诱导蛋白降解系统(Holland et al. 2012)来研究,以确定特定端粒酶成分的丢失是否会导致端粒酶RNA降解。回答端粒酶RNA寿命问题的工具也可以揭示端粒酶RNA整体生命周期的新细节。
如上所述,端粒酶rna是动态且不断变化的分子,在作用于端粒之前,要经历3 '和5 '端加工步骤,与转运蛋白结合,并与端粒酶组分组装。使用基于显微镜的方法,已经发现端粒酶rna定位在不同的细胞室中以经历这些成熟过程(Gallardo等人,2008,2011;Nguyen et al. 2015;Vasianovich et al. 2020;Buemi et al. 2022)。带有端粒酶RNA特异性荧光探针的FISH获得了有关RNA细胞区室分布的详细信息,并结合基于重组的分子工具,用于识别参与早期RNA穿梭的新组分(Zhu et al. 2004;Gallardo et al. 2008;Vasianovich et al. 2020)。利用免疫荧光技术已经实现了端粒酶RNA与蛋白质的定位,这有助于确定RNA何时被加工、穿梭或在端粒中起作用(Tomlinson等人,2006;Cristofari等人,2007;Cusanelli et al. 2013)。同样,特定端粒的荧光标记有助于确定细胞周期中端粒延伸的时间(Tomlinson et al. 2006)。使用活细胞方法更准确地记录了细胞中的端粒酶RNA动力学。对于人类和酵母端粒酶RNA, ms2标记RNA与GFP-MCP的偶联揭示了端粒酶RNA在细胞内的位置以及在细胞周期的不同阶段与端粒的关联,提供了端粒酶RNA整体生命周期的详细信息(Gallardo等人,2011;Laprade et al. 2020)。基于显微镜的研究端粒酶RNA动力学的方法是时间密集的,小规模的分析,需要专业知识和设备,而这些技术和设备并不广泛可用。然而,使用显微镜产生的视觉数据可以帮助解决有关端粒酶RNA定位,聚类和端粒作用的问题,这些问题可能不容易单独使用生化方法确定。事实上,了解端粒酶RNA加工的具体细节可以为疾病治疗提供途径。Cajal体中Tgs1对端粒酶RNA的5 '封顶(Verheggen et al. 2002)已被确定为潜在的治疗靶点,其中减轻Tgs1对端粒酶RNA的封顶并抑制端粒酶的作用可治疗癌症,或增加Tgs1端粒酶RNA的功能可用于治疗早衰疾病(Buemi et al. 2022)。
所有固定细胞和活细胞方法一起提供了端粒酶RNA生物发生的令人难以置信的细节,但仍有机会获得更好的分辨率。RNA标记的新工具和更高分辨率的显微镜,可以提供更详细的细胞过程观察继续发展。更亮的荧光蛋白(Lambert et al. 2020),直接结合RNA-荧光团的芒果和桃子RNA适配体(Cawte et al. 2020;Kong et al. 2021)和微流体装置(Gao et al. 2020;Anggraini et al. 2022),仅举几例,都可以提供更大更具体的端粒酶动力学细节,这是以前从未做过的。对于利用染料的新工具,以酵母为例,它需要一些优化,因为酵母细胞壁对外来分子是一个很大的屏障。虽然这一挑战可以通过突变多药转运泵来缓解,但HaloTag染料采用了一种成功的方法(Podh et al. 2022)。至于微流体设备,它们需要特殊和昂贵的设置,然而获得的长期成像细节是无与伦比的。微流体设备可以包括改变介质或添加药物、染料和营养素的选项,这些选项可以与诱导分子工具一起使用。显微镜的不断改进将使小细胞(在酵母的情况下)、细胞内室和长期成像的分辨率和清晰度更高,巩固了这些方法作为端粒酶研究的有力工具。
摘要。
介绍
端粒长度
端粒酶活性
端粒酶rna
端粒酶蛋白
端粒酶RNP的结构功能研究
超越实验室:计算机方法
结论
数据可用性
代码的可用性
参考文献。
致谢。
作者信息
道德声明
相关的内容
# # # # #
酵母和人类细胞中端粒酶的完整组成仍在研究中,因为它由许多不同的蛋白质组成,其中一些蛋白质只是端粒酶的一部分。在我们深入研究端粒酶蛋白分析方法之前,我们应该了解其中的关键角色。端粒酶最低限度地需要一种催化逆转录酶,在酵母中称为Est2,在人类中称为TERT,以及端粒酶RNA。在催化核心周围是一些与RNA支架或端粒酶亚基组装的蛋白质,其中一些对于端粒的亚细胞定位和结合是必要的。对于酵母,这些包括Est1, Est3, Pop1, Pop6, Pop7, yKu70/80二聚体和3 ' Sm7环(回顾见Bartle et al. 2022)。对于人类端粒酶,它们包括TCAB1、NHP2、GAR1、NOP10和dyskerin (Nguyen et al. 2018)。许多技术已被用于鉴定、表征和进一步研究与端粒酶形成相关和影响端粒酶形成以及随后与端粒相互作用的蛋白质。免疫共纯化蛋白已帮助鉴定酵母中的Est1、Est2、Est3、Pop1、Pop6、Pop7和Sm7蛋白,以及人类端粒酶RNPs中的TERT、NHP2、NOP10、dyskerin和GAR1蛋白(Harrington et al. 1997a, 1997b;Lingner et al. 1997a;Mitchell et al. 1999;Seto et al. 1999;Dragon et al. 2000;Zhou et al. 2000;Mitchell and Collins 2000;poga
伊奇等人,2000;Hughes et al. 2000;Fisher et al. 2004;Lemieux et al. 2016)。该方法依赖于感兴趣的表位标记蛋白,从粗提取物中纯化它们,然后通常使用逆转录酶定量PCR (RT-qPCR)或Northern blot来确定端粒酶RNA的存在。使用免疫沉淀,可以通过使用MS2-MCP系统间接下拉RNA来鉴定蛋白质(Laterreur等人,2018),通过质谱作为一种从头开始的方法来鉴定相关蛋白质(Lemieux等人,2016),并通过同时检测表位标记的蛋白质(共免疫沉淀;Witkin and Collins 2004;Fu and Collins 2007;Takai et al. 2010;Laterreur et al. 2018)。这些生化分析为端粒酶相关蛋白提供了强有力的证据,同时也与端粒酶活性分析相结合。
总的来说,上述方法产生了端粒酶RNPs的复杂细节,但没有一种方法接近低温电子显微镜(cryo-EM)获得的整体分辨率。最初,cryo-EM提供了纤毛端粒酶的三维结构(Jiang et al. 2015),揭示了催化核心内端粒酶RNA的新亚基和配置。最近,冷冻电镜技术被用于进一步定义人类端粒酶的3D结构,揭示亚纳米分辨率的端粒酶RNP构象(Nguyen et al. 2018;Liu et al. 2022)。这进一步确定了RNA和蛋白质亚基之间的相互作用位点,甚至揭示了端粒酶结构之谜的新片段。与人类端粒酶一样,我们预计酵母端粒酶与端粒酶RNA的3D结构也将通过低温电镜技术解决,因此可能提供有关该RNP的新细节。揭示端粒酶结构的复杂细节,为端粒延伸的拉链机制(Wan et al. 2021)和端粒酶相关疾病提供了更多细节。例如,TERT中的许多氨基酸突变导致疾病,通过冷冻电镜发现这些突变通常是TERT和端粒酶RNA之间的桥梁(Liu et al. 2022)。
低温电镜,不像其他蛋白质结构技术,不需要晶体。蛋白质复合体的多个角度的图像被合并,并使用专门的软件原子分辨率,创建蛋白质及其底物的3D模型(Weissenberger等人,2021)。尽管低温电镜技术可以获得难以置信的RNPs分辨率,但它代表了许多单个纯化配合物的平均结构。从冷冻电镜中获得结构的过程可能导致端粒酶RNA的少数形式(如3 '延伸和未加工形式)或与端粒酶RNP相互作用的瞬时蛋白被排除。区分断裂的rnp(由于方法过程)和不成熟的rnp也很困难。因此,仍然需要其他更具体的方法来评估端粒酶RNPs的全部种群。尽管如此,对端粒酶和端粒酶RNA结构的详细描述可以为端粒酶紊乱提供有价值的信息,这些紊乱是由端粒酶RNA和蛋白质突变引起的。
蛋白质纯化依赖的方法,如拉下试验或冷冻电镜已经非常有用的鉴定亚基组成端粒酶RNPs和研究端粒酶基因组位点的相互作用。然而,这些方法通常依赖于从细胞中大量提取蛋白质,不能提供亚细胞细节,可能导致代表性不足或无法检测瞬时蛋白质相互作用。将下拉分析或冷冻电镜与细胞分离相结合,可以提供室特异性端粒酶RNP组装或加工的更多具体细节,而单细胞分析可能有助于捕捉瞬时相互作用并可视化端粒酶的运动。
鉴于端粒酶是动态的,在细胞核和细胞质间穿梭,显微镜有助于我们了解端粒酶作用的时间。利用HaloTags,可以直接跟踪人类端粒酶,观察其何时作用于端粒,揭示了端粒酶-端粒结合的短关联和长关联实例,揭示了端粒酶-端粒结合的短暂性(Schmidt et al. 2016),以及端粒酶与端粒酶RNA的特定亚细胞定位和组装(Klump et al. 2023)。其他有前途的端粒酶显微镜工具是芒果和桃子RNA适配体(Cawte等人,2020;Kong et al. 2021)。荧光生物素与端粒酶RNA的直接结合为直接跟踪RNA和端粒酶提供了一种新的途径。此外,芒果和桃子的RNA适配体可用于蛋白质富集试验(Panchapakesan et al. 2017)。通过这种方式,实验可以使用相同的工具进行显微镜和蛋白质分析,因此实验之间存在直接关联。确定端粒酶在端粒中的结合时间和地点有助于提供有关端粒酶如何保留在端粒上以执行其功能的机制细节(Schmidt et al. 2018;Laprade et al. 2020)。我们目前对端粒酶动态性质的理解提出了端粒酶组分何时组装的问题。目前,尚不清楚每个蛋白质亚基何时在端粒酶RNA支架上聚集在一起,但利用免疫沉淀、生物素化和单细胞分析中的接近性测定可能为我们提供这些细节。关于不同细胞类型的端粒酶动力学,还有更多的东西需要学习,并且仍然有机会发现这种RNP在各种细胞和组织中的一生表现。
除了实验室技术外,还有计算机方法。高性能计算、基因组数据库、预测工具和专业软件的日益普及,将端粒研究领域推向了一个新时代。建模和其他复杂的统计分析在确定动力学和关联方面变得越来越有用,否则仅仅通过生化方法可能会错过。新的测序技术不仅使人们能够对表达变化有新的认识,而且还有助于预测蛋白质折叠(例如AlphaFold;Jumper等人,2021;Varadi et al. 2022),尤其是冷冻电镜后端粒酶结构的测定(Nguyen et al. 2018)。基于已知能量和碱基对之间吸引力的RNA折叠预测工具帮助科学家可视化RNA结构,最终帮助我们全面理解蛋白质-RNA相互作用(Zuker 1989, 2003;罗梅罗和布莱克本1991;Thompson et al. 1997;Chen et al. 2000;Tzfati et al. 2003;Dandjinou et al. 2004)。虽然由预测工具确定的结构可以,而且应该,通过基于实验室的方法进行测试和确认,因为硅折叠的参数很容易操作。
除了预测RNA和蛋白质结构工具之外,生物系统建模也有了很大的进步。复杂的统计、数学和生物学模型的结合,使人们对过去只能推测的细胞过程有了新的认识。生物系统的建模已被用于理解复制性衰老(Martin et al. 2021),产生的信息可能不仅仅来自实验室协议。建模也被证明对理解细胞中的端粒酶动力学非常有用(Laprade et al. 2020),并提供了一种从复杂和多因素数据中得出趋势的方法。除了建模之外,生物信息学的进步使大规模和复杂测序数据的分析成为可能(Sholes等人,2022;Tham et al. 2023;D 'Angiolo et al. 2023),利用高性能计算和创建特定的软件或生物信息学工具来满足我们研究问题的需求。
随着实验室和计算机科学的不断进步,将会开发出一些伟大的工具,使下一代科学家能够以以前的研究人员甚至无法想象的方式,更深入地研究端粒酶的结构、功能和生物发生。
广泛的生物化学和计算机技术使我们对端粒酶的复杂特性和动力学有了目前的了解。虽然端粒酶活性和端粒长度测定等金标准方法仍然是实验中初始表型确认的有用工具,但使用最新一代技术,如长读测序,冷冻电镜和建模,已经导致对这一重要RNP的结构和功能的更精确和更详细的看法。尽管我们有比以往更多的技术来研究端粒酶,但当涉及到研究端粒和/或端粒酶RNP时,人类细胞系和种群研究遇到了特殊的问题。人类细胞系的长期分析仅限于永生化细胞类型,这些细胞类型可能显示出由细胞问题引起的端粒表型,因此可能无法反映正常细胞中会发生的情况。在更大的范围内,人类研究依赖于血液样本来研究人群的端粒,因此会错过其他组织特有的端粒或RNP变化的趋势。这突出表明,在人类的生命周期中,从人口规模到单个细胞,关于端粒和端粒酶RNP还有很多需要学习的地方。
需要新的技术和程序来回答端粒酶和端粒尚未解决的问题(图1),包括3 '端粒悬垂是否与健康和疾病有关;端粒酶RNA是如何降解的;端粒酶蛋白何时何地聚集;端粒酶RNA波动发生了吗?它是如何被调节的?蛋白质和RNA修饰在疾病和一般健康中起作用吗?端粒酶rna的进化如何影响生物体的成功,为什么端粒酶rna在不同的进化枝上如此不同?端粒酶RNA结构的这些差异是否与端粒酶RNA的有效性有关?端粒酶在端粒维护之外是否还有作用?它涉及到什么分子机制或生物学意义?既然我们知道细胞间室在生物化学上不是均匀的,那么细胞核和细胞质的组织和结构是如何影响端粒酶的组装、运动和功能的呢?
随着精确度和能力的提高,新技术可以帮助回答这些问题,并继续揭示端粒酶的复杂机制。
ccDownload: /内容/ pdf / 10.1007 / s10522 - 023 - 10073 - 8. - pdf



